1.SegCLR:対照学習で教師なしで人間の脳をマッピングを作成(1/2)まとめ
・人間の脳の配線や情報伝達経路をマッピングすること重要な基礎研究分野
・脳は大量の情報を持つ複雑な構造のため学習用のラベル付けが困難な領域
・SegCLRを使うと細胞の形状や超微細構造を手間をかけずに学習可能となる
2.SegCLRとは?
以下、ai.googleblog.comより「Multi-layered Mapping of Brain Tissue via Segmentation Guided Contrastive Learning」の意訳です。元記事の投稿は2022年11月9日、Peter H. LiさんとSven Dorkenwaldさんによる投稿です。
アイキャッチ画像はstable diffusionの1.5版の生成で脳とトトロのつもりだったのですが、Brainが中々脳になってくれず、ブレーン(参謀)の意味なのかマッドサイエンティスト風やブラックジャック風になってしまうトトロ
人間の脳の配線や発火活動をマッピングすることは、私たちがどのように考え、世界を感じ、学習し、決定し、記憶し、創造するか、また、脳の病気や機能障害でどのような問題が生じるかを解読するための基礎となります。
最近の取り組みにより、これまでにない品質と規模の脳マップ(脳細胞とその結合の高解像度3Dマッピング)が一般に公開されるようになりました。
例えば、H01はハーバード大学とグーグル社による人間の脳組織をナノメートル単位でデジタル再構成した1.4ペタバイトサイズのデータであり、MICrONSコンソーシアムの同僚たちはミリメートル立法単位のマウスの大脳皮質データセットを提供しています。
この規模の脳マップを解釈するためには、シナプス結合、細胞サブコンパートメント、細胞タイプの特定など、何層もの分析が必要です。機械学習やコンピュータビジョン技術は、こうした解析を可能にする中心的な役割を担っていますが、こうしたシステムの導入には、専門の注釈付け作業者による何時間もの手作業の検証済ラベルの付与作業と、膨大な計算資源が必要であり、依然として手間のかかるプロセスとなっています。さらに、軸索や樹状突起の小さな断片から細胞の種類を特定するような重要なタスクは、人間の専門家にとっても困難であり、効果的な自動化はまだ行われていません。
本日、「Multi-Layered Maps of Neuropil with Segmentation-Guided Contrastive Learning」において、細胞の形状(cellular morphology)と細胞の超微細構造(ultrastructure)の豊かで汎用的な表現を、手間をかけずに学習する手法、SegCLR(Segmentation-Guided Contrastive Learning of Representations)を発表します。
SegCLRはコンパクトなベクトル特徴表現(embeddings)を生成し、様々な下流タスク(例:細胞の一区画を局所的に分類、教師なしクラスタリング)に適用でき、細胞の小さな断片から細胞の種類を識別することも可能です。
私達は、H01ヒト大脳皮質データセットとMICrONSマウス大脳皮質データセットの両方でSegCLRを訓練し、得られたembeddingベクトル(合計約80億)を研究者に公開しています。
SegCLRのembeddingは、3Dブロックから切り出された脳細胞から、細胞の形態と超微細構造を捉え、細胞の一区画(例:樹状突起スパインと樹状突起シャフト)や細胞タイプ(例:錐体細胞とミクログリア細胞)を区別するために使用することができます。
細胞の形態と超微細構造の特徴表現
SegCLRは、自己教師あり対照学習における最近の進歩に基づいて構築されています。標準的なディープネットワークアーキテクチャを用い、電子顕微鏡データの局所的な3Dブロック(一辺が約4マイクロメートル)からなる入力を64次元のembeddingベクトルにエンコードします。
ネットワークは、意味的に関連する入力をembedding空間内の類似した座標にマッピングするために、対照的損失を通じて学習します。これは一般的なSimCLRの設定に近いです。ただし、ボリュームのインスタンスセグメンテーション(個々の細胞や細胞片をトレースする)も必要で、これを2つの重要な方法で利用します。
まず、入力された3次元電子顕微鏡データはセグメンテーションによって明示的にマスクされ、ネットワークは各ブロック内の中心細胞のみにフォーカスするように強制されます。
第二に、セグメンテーションを利用して、どの入力が意味的に関連しているかを自動的に定義します。対照損失では、正のペアはセグメンテーションされた同じ細胞の近距離から抽出され、類似した特徴表現を持つように訓練されます。異なる細胞からの入力は、異なる特徴表現を持つように訓練されます。
重要なのは、一般に公開されているヒトとマウスのデータセットを自動でセグメンテーションした結果は、人間の専門家による面倒なレビューや修正を必要とせずに、SegCLRを訓練するのに十分な精度であったということです。
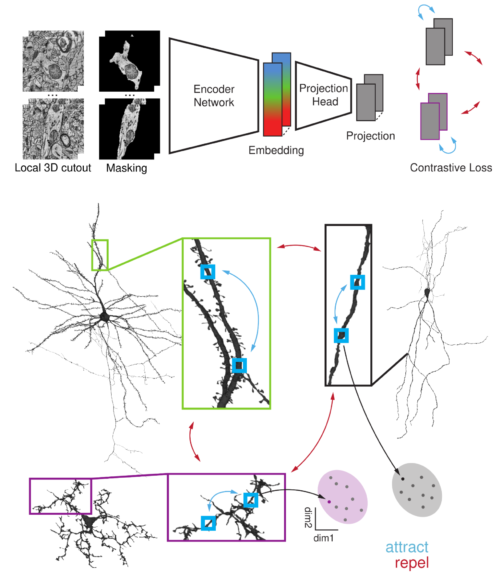
SegCLRは、手動でラベル付けすることなく、豊富な細胞特徴を表現するように訓練されています。上:SegCLRは、電子顕微鏡データの局所的にマスクされた3Dビューをembeddingベクトルにマッピングするアーキテクチャです。必要なのは、顕微鏡像のボリュームと、自動インスタンスセグメンテーションの下書きのみです。下図:セグメンテーションは、ポジティブな例とネガティブな例のペアを定義するためにも使用され、学習中にその特徴表現が近づけられます(似た事例同士、青い矢印)、遠ざけられる(異なった事例同士、赤い矢印)。
学習時の注釈の必要性を3桁削減
SegCLRのembeddingは、教師あり(分類器の学習など)、教師なし(クラスタリングやコンテンツベースの画像検索など)を問わず、様々な下流工程で利用することが可能です。教師ありの場合、embeddingは分類器の学習を簡素化し、グランドトゥルースラベリングの必要性を大幅に削減することができます。例えば、細胞の一区画(軸索、樹状突起、体細胞など)を識別する場合、SegCLRのembeddingの上で学習した線形分類器は、同じタスクで学習した完全教師ありディープネットワークを上回り、しかも数百万のラベル付きサンプルではなく、千程度のサンプルしか使わないことを発見しました。
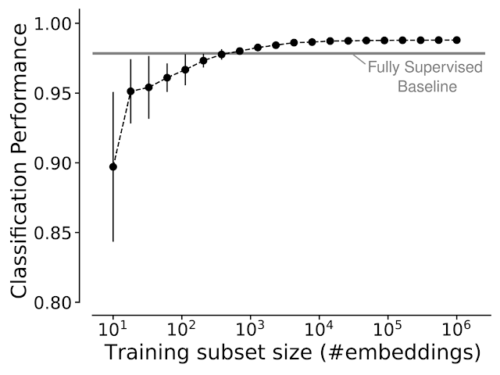
私達は、使用するトレーニング事例の数を変えながら、平均F1-Scoreを介して、ヒト皮質データセットの軸索、樹状突起、ソーマ、およびアストロサイトの一区画の分類性能を評価しました。SegCLRのembeddingの上で学習した線形分類器は、学習データの一部を使用しながら、完全教師ありディープ分類器(水平線)の性能と一致するか、それを上回りました。
小さな断片からでも、細胞の種類を見分ける事が可能
異なる種類の細胞を識別することは、健康や病気において脳回路がどのように発達し、機能するかを理解するための重要なステップです。人間の専門家は、形態学的特徴に基づいて皮質細胞の種類を識別できるようになりますが、手作業による細胞種識別は手間がかかり、あいまいなケースもよくあります。また、細胞の小さな断片しか得られない場合にも、細胞種識別は難しくなります。
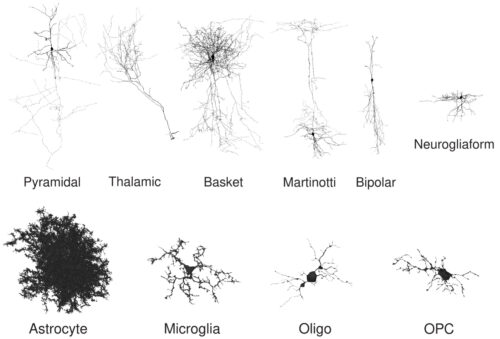
各データセットにおいて、少数の確認済み細胞に対して、人間の専門家が手作業で細胞タイプをラベル付けしました。マウス大脳皮質データセットでは、専門家が6つのニューロンタイプ(上)と4つのグリアタイプ(図示されてません)をラベル付けしました。ヒト大脳皮質データセットでは、専門家が2つのニューロンタイプ(図示されてません)と4つのグリアタイプ(下)をラベル付けしました。(行中の縮尺は変えていません)。
3.SegCLR:対照学習で教師なしで人間の脳をマッピングを作成(1/2)関連リンク
1)ai.googleblog.com
Multi-layered Mapping of Brain Tissue via Segmentation Guided Contrastive Learning
2)www.biorxiv.org
Multi-Layered Maps of Neuropil with Segmentation-Guided Contrastive Learning
3)h01-release.storage.googleapis.com
SegCLR Embeddings