1.SegCLR:対照学習で教師なしで人間の脳をマッピングを作成(2/2)まとめ
・SegCLRは脳細胞の小さな断片に対してもヒトとマウスの細胞型を正確推定可能
・マウス大脳のシナプスの接続相手を分類する事で脳結合性の自動解析も可能
・リッチでコンパクトなembedding表現により生物学的な洞察の新しい道を開く
2.SegCLRの性能
以下、ai.googleblog.comより「Multi-layered Mapping of Brain Tissue via Segmentation Guided Contrastive Learning」の意訳です。元記事の投稿は2022年11月9日、Peter H. LiさんとSven Dorkenwaldさんによる投稿です。
アイキャッチ画像はstable diffusionの1.5版の生成
SegCLRは、小さな断片に対しても、ヒトとマウスの細胞型を正確に推定することがわかりました。分類の前に、中心点からの半径として定義される凝集距離にわたって、各細胞内の埋め込みを収集し、平均化しました。
その結果、ヒトの皮質細胞は、ミクログリア(MGC:microglia)とオリゴデンドロサイト前駆細胞(OPC:oligodendrocyte precursor cells)のような専門家が区別しにくいタイプであっても、10マイクロメートルという小さな凝集半径で高い精度で同定できることがわかりました。
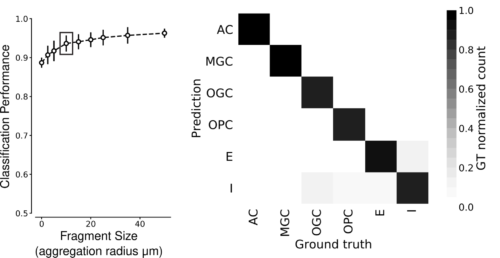
SegCLRは小さな断片からでも、細胞の種類を分類できます。
左:異なるサイズの細胞断片に対してSegCLRの埋め込みで学習した浅いResNetモデルの分類性能(6つのヒト大脳皮質の細胞タイプを分類)。集計半径0は、embeddingが1つしかない非常に小さな断片に対応します。細胞タイプの分類性能は、凝集半径がわずか10マイクロメートルの断片に対して高い精度(0.938平均F1-スコア)に達しています(枠で囲んだ点)。
右:集計半径10μmでのクラスごとの混同行列。対角線に沿った濃い網掛けは、予測された細胞タイプがほとんどのケースで専門家のラベルと一致することを示しています。AC:アストロサイト、MGC:ミクログリア細胞、OGC:オリゴデンドロサイト細胞、OPC:オリゴデンドロサイト前駆細胞、E:興奮性神経細胞、I:抑制性神経細胞。
マウス大脳皮質では、25マイクロメートルの集計半径で10種類の細胞が高い精度で識別できました。
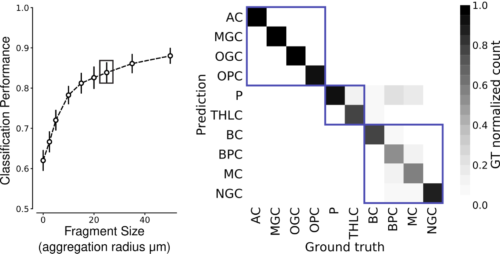
左:マウス大脳皮質の10種類の細胞について、集計半径25μmの断片の平均F1-Scoreは0.832に達しました(枠で囲んだ点)。
右:集計半径25ミクロンでのクラスごとの混同行列。箱は大分類(グリア、興奮性ニューロン、抑制性介在ニューロン)を示します。P:錐体細胞、THLC:視床皮質軸索、BC:バスケット細胞、BPC:双極細胞、MC:マルティノッティ細胞、NGC:神経膠質細胞。
さらに細胞タイプのアプリケーションでは、SegCLR embeddingを使用した教師なしクラスタリングを使用して、さらに神経細胞のサブタイプを明らかにし、不確実性推定を使用して、データセットの信頼性の高いサブセットに分類を制限できることを実証しました。(例えば、少数の細胞タイプのみが専門家のラベルを持つ場合など)
脳内結合のパターンを明らかにする
最後に、私達はSegCLRが、マウス大脳皮質データセット全体を通して再構成されたシナプスの接続相手を分類することによって、脳結合性の自動解析にどのように使用できるかを示しました。
特定の細胞タイプ間の結合パターンを知ることは、大規模な脳配線再構成を解釈する上で基本的なことですが、これには通常、接続先となる細胞種を特定するための手作業によるトレースが必要です。
SegCLRを用いることで、これまで手作業によるトレースに頼っていた脳結合の知見を再現するとともに、シナプス数、細胞種、解析脳領域などの規模を拡大することができました。(詳しくは論文をご覧ください)
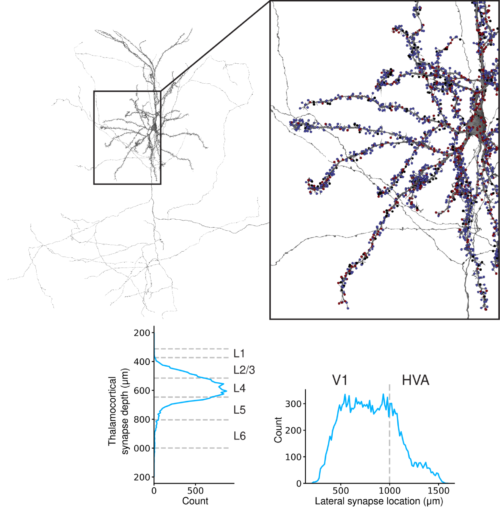
SegCLRによる脳内結合の自動解析
上図:マウスの錐体細胞の例。シナプスの位置は、シナプスの相手が抑制性(青)、興奮性(赤)、不明(黒)に分類されているかどうかによって色分けされています。挿入図は、ソーマと近位樹状突起の詳細を示します。
下図:シナプス上流のうち、感覚系から大脳皮質に入力をもたらす視床皮質軸索に分類されるものがどれだけあるかを数えました。視床からの入力は、主に皮質の標準的な入力層であるL4層に到達し、高次視覚野(HVA)よりもむしろ一次視覚野V1を優先的にターゲットにしていることがわかりました。
次のステップは?
SegCLRは豊富な細胞の特徴を捉え、生の画像やセグメンテーションデータを直接扱うよりも下流の分析を大幅に簡素化することができます。
ヒトとマウスの皮質データセットについて、約80億のembeddingをリリースしています。(Neuroglancerを使ってヒトとマウスの脳再構築をブラウザで閲覧可能)。
SegCLRは、複雑な顕微鏡データをリッチでコンパクトなembedding表現にすることで、生物学的な洞察のための多くの新しい道を開き、細胞や細胞区画レベルでの高次元の特徴づけのための補完的な様式、例えば空間分解トランスクリプトーム(spatially-resolved transcriptomics)へのリンクとして機能する可能性があります。
3.SegCLR:対照学習で教師なしで人間の脳をマッピングを作成(2/2)関連リンク
1)ai.googleblog.com
Multi-layered Mapping of Brain Tissue via Segmentation Guided Contrastive Learning
2)www.biorxiv.org
Multi-Layered Maps of Neuropil with Segmentation-Guided Contrastive Learning
3)h01-release.storage.googleapis.com
SegCLR Embeddings