1.機械学習を使ってゲノムの発見を改善(2/2)まとめ
・機械学習を使った手法と人間の専門家が手動で全画像を診断した手法を比較した
・機械学習は専門家が見つけた65の関連のうち62を見付け更に93の新規関連を発見
・機械学習が発見した新しい関連を応用して緑内障の兆候も高い精度で予測できた
2.機械学習を使ったGWASの改善
以下、ai.googleblog.comより「Improving Genomic Discovery with Machine Learning」の意訳です。元記事の投稿は2021年6月23日、 Andrew CarrollさんとCory McLeanさんによる投稿です。
アイキャッチ画像のクレジットはPhoto by Catrina Carrigan on Unsplash
MLベースのGWASは、VCDRに関連する156の異なるゲノム領域を特定しました。これらの結果を、同じUK Biobankデータで別のグループ(Craig et al. 2020)が行った結果と比較しました。この実験では専門家がVCDRのすべての画像に丹念にラベルを付けています。
MLベースのGWASはCraig et alで見つかった65の関連のうち62を見付けました。これは、モデルがUK Biobank画像のVCDRを正確に予測していることを示しています。さらに、MLベースのGWASは、93の新規関連を発見しました。
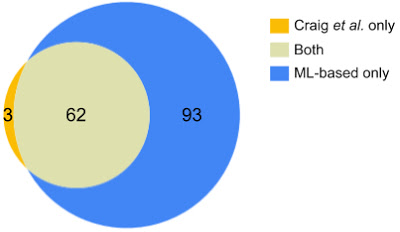
徹底的なエキスパートラベリングアプローチ(Craig et al.左)とMLベースのアプローチ(右)によって発見された統計的に有意なGWASアソシエーションの数。中央のアソシエーションは両者で見つかったものです。
MLベースのGWASによるポリジーンモデルの予測改善
MLベースのGWASで発見された新しい関連性が生物学的に関連していることを検証するために、Craig et alとMLベースのGWAS結果を使用して独立したPRSを開発しました。
そして、UK Biobankデータセットと完全に異なる独立した集団(EPIC-Norfolk)に対して、人間の専門家がラベルを付けたVCDRを予測する能力をテストしました。
MLベースのGWASから開発されたPRSは、両方のデータセットでエキスパートラベリングアプローチから構築されたPRSよりも優れた予測能力を示し、MLベースの方法によって発見された新しい関連性がVCDR生物学に影響を与えるという強力な証拠を提供します。
そして、モデルの表現型の精度の向上(つまり、より正確なVCDR測定)がより強力なGWASにつながることを示唆しています。
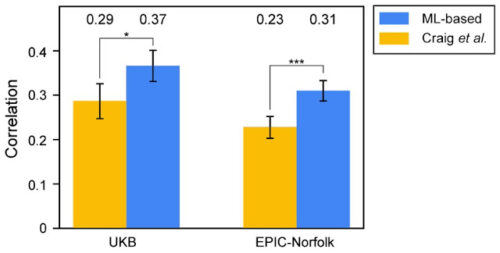
MLベースのアプローチから生成されたVCDRのポリジーンリスクスコア(PRS)と徹底的に専門家がラベリングしたアプローチ(Craig et al.)の相関関係。これらのグラフでは、y軸の値が高いほど相関が大きく、したがって遺伝データのみからの予測が出来ていることを示します。 [* p ≤ 0.05; *** p ≤ 0.001]
2番目の検証として、VCDRは緑内障と強く相関していることがわかっているため、MLベースのPRSが、緑内障であると自己申告した個人、または緑内障または緑内障治療を示唆する医療処置コードを持っていた個人と相関しているかどうかを調査しました。
モデル予測を使用して決定されたVCDRのPRSは、個人が緑内障の兆候を示している確率も予測できることがわかりました。PRSの標準偏差が2.5以上平均よりも高い個人は、この集団内で緑内障を発症する可能性が3倍以上でした。また、MLベースの表現型からのVCDR PRSは、広範な手動表現型から生成されたVCD RPRSよりも緑内障を予測しやすいことも観察されました。
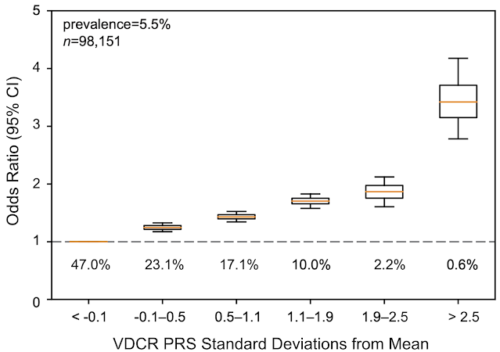
MLベースの表現型を使用して決定されたVCDRのPRSによって層別化された緑内障(自己報告またはICDコード)のオッズ比(平均からの標準偏差)。このグラフでは、y軸は、ベースライン率(破線)と比較して、個人が緑内障を患っている確率を示しています。 x軸は、PRSの平均からの標準偏差を示しています。データは、平均(オレンジ)、第1四分位数と第3四分位数、および最小値と最大値の値を示す標準の箱ひげ図として視覚化されます。
結論
MLモデルを使用して、GWASで大規模集団の表現型分析ををすばやくできること、およびこれらのモデルを使用して、このような研究の統計的検出力を高めることができることを示しました。これらの例は、網膜イメージングから予測された目の特徴について示されていますが、この概念が他の疾患やデータタイプに一般的にどのように適用できるかを探求することを楽しみにしています。
謝辞
特に、ムーアフィールズ眼科病院の共著者であるDr. Anthony Khawajaが幅広い医学的専門知識で貢献してくれたことに感謝します。また、UK Biobank画像の徹底的なラベリングを行ったJamie Craig教授とその同僚の努力にも感謝します。これにより、私たちの方法と比較することができました。 その研究を行った何人かの著者、ならびにStuart MacGregor 教授、オーストラリアの共同研究者とMax Kelsen社は、これらの発見を独自に複製しており、私たちはこれらの科学的貢献も高く評価しています。
最後に、この研究は、次のGoogleの貢献者の作品をまとめたもので感謝の意を表したいと思います。Babak Alipanahi, Farhad Hormozdiari, Babak Behsaz, Justin Cosentino, Zachary R. McCaw, Emanuel Schorsch, D. Sculley, Elizabeth H. Dorfman, Sonia Phene, Naama Hammel, Andrew Carroll, そして Cory Y. McLean。
3.機械学習を使ってゲノムの発見を改善(2/2)関連リンク
1)ai.googleblog.com
Improving Genomic Discovery with Machine Learning
2)www.cell.com
Large-scale machine-learning-based phenotyping significantly improves genomic discovery for optic nerve head morphology
3)github.com
Google-Health / genomics-research / ml-based-vcdr /