1.DeepVariant 1.0によるゲノム解析精度の向上(4/4)まとめ
・PrecisionFDA前はエラー率が高いため、Oxford Nanoporeデータ用は使用できなかった
・UC Santa Cruz Genomics Instituteと提携し彼らの持つPEPPERを汲み込んだ
・PEPPER-DeepVariantはOxford NanoporeでIlluminaよりも優れた精度を達成
2.PEPPER-Deepvariant
以下、ai.googleblog.comより「Improving the Accuracy of Genomic Analysis with DeepVariant 1.0」の意訳です。元記事の投稿は2020年9月18日、Andrew CarrollさんとPi-Chuan Changさんによる投稿です。
アイキャッチ画像のクレジットはPhoto by Nicolas Hoizey on Unsplash
(5)リリースしたモデルを更にトレーニングするために学習データを追加
コンテストの時間枠が短縮されたため、モデルのトレーニングが高速化できるように、コンテスト用データ(PCR-Free NovaSeq)と似たデータのみを使ってトレーニングしました。DeepVariant v1.0の製品リリースでは、PCR+だけでなく、複数の機器にも高い精度を求めています。これらの多様なデータを使用したトレーニングは、モデルの一般化に役立ちます。そのため、DeepVariant v1.0のリリースモデルは、コンテストに提出されたモデルよりも優れています。
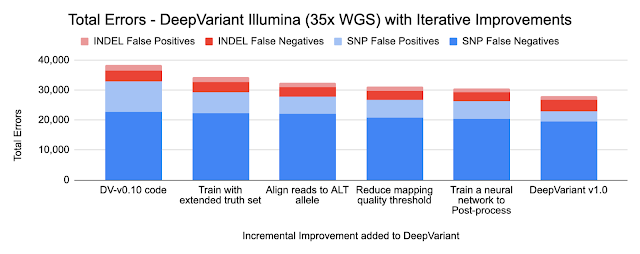
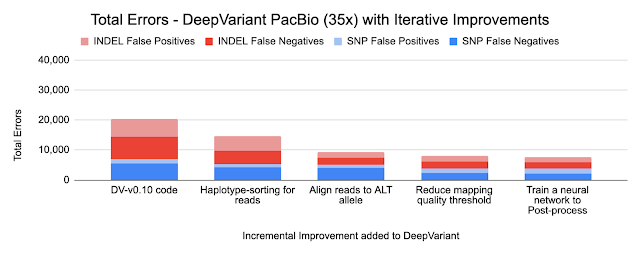
以下のグラフは、各改善によって達成されたエラーの削減を示しています。2種類の小さなvariants、SNP(Single Nucleotide Polymorphisms、一塩基多型、配列の長さを変更せずに単一の塩基を変更)とINDEL(挿入と削除)のIlluminaとPacBioの数値を示します。
ハイブリッドモデルのトレーニング
DeepVariant v1.0には、PacBioとIlluminaの2つの読み取りに対応可能なハイブリッドモデルも含まれています。この場合、モデルは新しいロジックを必要とせずに、両方の入力タイプの長所を活用します。

PacBioとIlluminaの両方からのデータを併合するDeepVariantの例
6つのデータチャネルのうち2つだけが示されています。チャネル#1「Read base」とチャネル#5「Read supports variant」です。長いPacBioの読み取り(画像上部)は完全に呼び出し領域に展開されますが、短いIlluminの読み取りは領域の一部のみに展開されます。
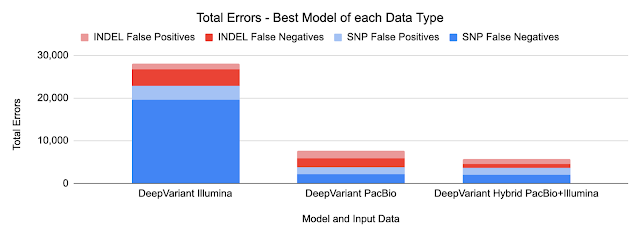
SNPエラーに変化は見られなかったことから、PacBioの読み取りはSNP呼び出しよりも厳密に優れていることがわかります。
私達はPacBioモデルと比較して、Indelエラー(挿入欠失エラー)がさらに49%減少する事を観察しました。これは、IlluminaとPacBio HiFiのIndelエラーモードを補完的に使用できることを示唆しています。
PEPPER-Deepvariant:DeepVariantを使用したOxford Nanoporeデータのパイプライン
PrecisionFDAのコンテスト開催前は、ベースエラー率が高く、DeepVariantが分類できない候補が多すぎるため、Oxford Nanoporeデータ用にはDeepVariantモデルは利用できませんでした。
私達は、Oxford Nanoporeのデータに関する豊富な専門知識を持つUC Santa Cruz Genomics Instituteと提携しました。
彼らは以前、PEPPERと呼ばれるディープラーニング手法をトレーニングしていました。これを使うと候補をより扱いやすい数に絞り込むことができます。DeepVariantのより大きなニューラルネットワークは、妥当な実行時間で残りの候補を正確に特徴付けることができます。
PEPPER-DeepVariantパイプラインとOxford Nanoporeモデルを組み合わせたものはオープンソースとして公開されており、GitHubで入手できます。このパイプラインは、PrecisionFDAコンテストでDeepVariant Illuminaよりも優れたSNP呼び出し精度を達成することができました。このようにしてOxford NanoporeがIlluminaよりも優れていることを示したのは初めての事です。
結論
DeepVariant v1.0は開発の終了を意味するわけではありません。ゲノミクスコミュニティと協力して、患者や研究者にとってのゲノムデータの価値をさらに最大化できることを楽しみにしています。
3.DeepVariant 1.0によるゲノム解析精度の向上(4/4)関連リンク
1)ai.googleblog.com
Improving the Accuracy of Genomic Analysis with DeepVariant 1.0
2)www.nature.com
A universal SNP and small-indel variant caller using deep neural networks
3)github.com
google / deepvariant
kishwarshafin / pepper
4)www.nist.gov
Genome in a Bottle