1.Chemome initiative:機械学習を使って有用なタンパク質を探る試み(1/2)まとめ
・人間の病気の治療薬は、タンパク質の働きを調整する目的で投与される事が多い
・小分子薬はタンパク質の機能や相互作用を阻害または促進することによって機能する薬
・タンパク質の機能を調べる手法を機械学習でもっと効率化できないだろうか?
2.Chemome initiativeとは?
以下、ai.googleblog.comより「Unlocking the “Chemome” with DNA-Encoded Chemistry and Machine Learning」の意訳です。元記事の投稿は2020年6月11日、Patrick Rileyさんによる投稿です。
有用なタンパク質を発見する作業は新薬開発にとって非常に重要なのですが、従来は熟練技術者が人海戦術で行う側面があり、以前、画像認識人工知能を使ってタンパク質の結晶を発見する話を訳した事があります。
今回は、タンパク質のDNAを元に似たような性質を持つタンパク質を推論しようとするパターン認識の問題です。
アイキャッチ画像のクレジットはアメリカ国立がん研究所提供画像で資料をチェックするアフリカ系アメリカ人の男性研究員と女性研究員
人間の病気の治療薬は、タンパク質の働きを調整する目的で投与される事が多いです。タンパク質は多くの生物学的活動の主要な担い手であるためです。
イブプロフェンなどの小分子薬(Small molecule drugs)は、タンパク質の機能またはタンパク質と他の生体分子との相互作用を阻害または促進することによって機能するタイプが多いです。
潜在的に有用な小分子を、実験室の実験ではなく、何らかの計算で見つける事ができる「仮想スクリーニング(virtual screening)」法の開発は、長い間の研究課題でした。
しかしながら、より求められている課題は、「対象タンパク質との間に物理的に検証された有用な相互作用」つまり「ヒット(hits)」を持つ小分子を見つける手法です。幅広い化学分野にわたって十分に機能するヒット探索手法が求められています。
Journal of Medicinal Chemistryで最近発表された論文「Machine learning on DNA-encoded libraries: A new paradigm for hit-finding」では、X-Chem Pharmaceuticalsと協力して、生物学的に効力がある分子を見つける効果的な新しい手法を示しました。
「DNAをエンコードした小分子ライブラリを使った物理スクリーニング」と「グラフ畳み込みニューラルネットワーク(GCNN)を使用した仮想スクリーニング」の組み合わせを使用します。
この研究は、「Chemome initiative(ケモム構想)」の創設につながりました。これは、Google Accelerated Science teamとZebiAIの間の共同プロジェクトであり、生物学的研究のためにより多くの有用な小分子の働きを探査する事を可能にします。
化学的探査装置の背景
生命を支え、病気を生み出す生物学的ネットワークを理解することは、非常に複雑な作業です。
これらのプロセスを研究する1つのアプローチは、「化学的探査装置(chemical probes)」を使用することです。化学的探査装置とは、薬物として必ずしも有用ではないが、特定のタンパク質の機能を選択的に阻害または促進する小分子です。
研究対象の生物系(実験室のガラス皿で増殖するがん細胞など)がある場合、特定の時間に化学的探査装置を追加します。これにより、標的タンパク質の活性が増加または減少するなど、生物系の反応を観察する事ができます。
しかし、基本的な生物医学研究にこの種の化学的探査装置が非常に有用であるにも関わらず、人間のタンパク質の4%だけが既知の化学的探査装置を利用できます。
化学的探査装置を見つけるプロセスは、小分子薬を見つける初期段階と同様です。
目的のタンパク質に対して、膨大な小分子の集まり中から、「ヒット」を持つ分子を見つけます。
数十万または数百万もの分子を物理的に試験できる「ロボットを支援に使った効率のよいスクリーニング」手法は、現代の薬物研究の基礎です。
ただし、簡単に購入できる小分子の数(1.2 × 10の9乗)はロボット支援スクリーニングが扱える数よりはるかに多いです。それでも、分子のような小分子薬の数(10の20乗から60乗と推定)よりははるかに少ないのです。
「仮想スクリーニング」手法は、潜在的に合成可能な分子の莫大な組み合わせを迅速かつ効率的に検索する事で、治療用化合物の発見を大幅にスピードアップする可能性があります。
DNAをエンコードした小分子ライブラリのスクリーニング
スクリーニングプロセスの物理的な部分では、DNAをエンコードした小分子ライブラリ(DEL:DNA-encoded small molecule libraries)を使用します。
DELには1つのプールに多数の異なる小分子が含まれています。それぞれがその分子の一意のバーコードとして機能するDNAの断片に関連付けられます。
この基本的な手法は数十年前から存在していますが、ライブラリとスクリーニングプロセスの質は、有意義な結果を生み出すための鍵となります。
DELは、生化学的課題を解決するための非常に賢いアイデアです。DELにより、小さな分子を1つの場所に集めて、それぞれを簡単に識別する事ができます。
重要な事は、ノーベル賞を受賞したファージディスプレイ技術(phage display technology)と同様に、DNAをバーコードとして使用して各分子を識別することです。
まず、沢山の化学的フラグメント(Chemical fragments)を生成します。化学的フラグメントとは、化学構造の基礎的土台であり、これを使って化学物質の特性をモデリングする際に使うと直感的に理解しやすくなります。
各化学的フラグメントに、それぞれに固有のDNAバーコードを付け、共通の化学ハンドル(下図ではNH2)を付けます。次に、反応毎に分割し、個々の化学フラグメントに別の化学ハンドル(OHなど)を追加します。
2つのステップにより、化学的フラグメントは、共通の化学ハンドルを使って表現されます。DNAフラグメントも接続され、各分子に対して1つの連続したバーコードが作成されます。
最終的に、2N回の操作を実行することにより、Nの二乗個の一意の分子を取得し、それぞれが独自の一意のDNAバーコードによって識別できるようになります。より多くのフラグメントまたはより多くこの作業を繰り返す事により、数百万または数十億もの異なる分子を含むライブラリーを比較的簡単に作成できます。
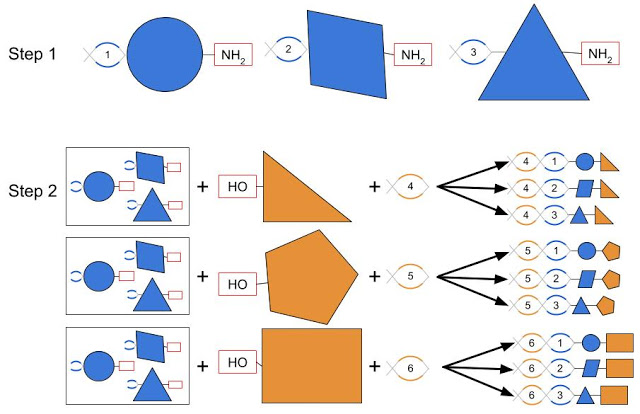
DNAエンコードした小分子ライブラリを作成するプロセスの概要
まず、小さな化学フラグメント(青で表現)にDNA「バーコード」(ここでは番号の付いた螺旋で表現されています)が、共通の化学「ハンドル」(上図のNH2など)を明確にするために取り付けられます。
他の化学フラグメント(オレンジで表現され別の化学「ハンドル」(HO)を持ちます)と混合した時、科学反応により、化学フラグメントとDNAフラグメントのセットが併合され、それぞれ固有のDNA「バーコード」を持つ、小分子の膨大なライブラリーが生成されます。
ライブラリーが完成したら、DELをタンパク質と混合し、付着していない小分子を洗い流すことにより、目的のタンパク質に結合する小分子を見つけるために使用できます。
残りのDNAバーコードの結合順序を決定すると、何百万ものDNAフラグメントが読み取りできます。これを注意深く処理して、元のDELの何十億もの分子がタンパク質と相互作用するか否かを推定できます。
3.Chemome initiative:機械学習を使って有用なタンパク質を探る試み(1/2)関連リンク
1)ai.googleblog.com
Unlocking the “Chemome” with DNA-Encoded Chemistry and Machine Learning
2)pubs.acs.org
Machine Learning on DNA-Encoded Libraries: A New Paradigm for Hit Finding



コメント