1.GWASkb:ゲノムワイド関連解析情報を論文から自動抽出(4/6)まとめ
・GWASkbは多くのアプリケーションに有用なレベルで手動キュレーションDBに掲載された8割を自動収集
・GWASkb手動キュレーションDBに掲載されていないが有用であると思われる多様体も収集できていた
・多様体を外部ツールによる注釈と比較した所、有意に一致しており外部ツールによる検証もクリア
2.GWASkbと他のGWAS関連データベースとの比較
以下、www.nature.comより「A machine-compiled database of genome-wide association studies」の意訳です。元記事は2019年7月26日、Volodymyr Kuleshovさん、Jialin Dingさん、Christopher Voさん、Braden Hancockさん、Alexander Ratnerさん、Yang Liさん、Christopher Réさん、Serafim BatzoglouさんとMichael Snyderさんによる投稿です。
GWASkbで抽出できたデータセットには、ほぼ正しい表現型を持つGWAS Centralからの2487(82%)の関連性と、GWAS Catalogからの3245(81%)の関連性が含まれていました。
また、完全な正確さで抽出できた表現型は、GWAS Centralから1890(63%)、GWAS Catalogから2762(69%)の関連性を回復できています。GWASkbの表現型が間違っており、一部の関連付けが正しく復元されなかった割合はGWAS Centralで89(3%)およびGWAS Catalogで147(4%)です。
残りのケースでは、多様体自体を報告できませんでした。これの主な原因は、多様体がテーブルではなくテキストのみで表現されている場合、またはテーブルの形式の解析が特に困難な場合(例えば、複数のRSIDとp値が同じ行で報告される場合など)です。 全体として、GWASkbは、多くのアプリケーションで役立つ品質レベルで、手動でキュレーションされた関連付けの81 – 82%を回復しました。
機械による自動キュレーションは有用な新しい関連性を発見しました
合計で、GWASkbには589の入力論文内に6422の関連性が含まれ、そのうち2959(46%)はGWAS Catalog内の情報にもGWASセントラル内の情報にもマッピングできませんでした。
最初にランダムに抜き出した100の新しい関連性を検査することで(2名の独立した注釈者が別々に検証する事で)調査しました。
新たに発見された100の関連性のうち、88の関連性はシステムの仕様を完全に満たしており、7つの関連性は間違っており、5つの関連性が別の研究によって最初に特定された(そして背景資料として論文内で参照されていた)事がわかりました。私達のシステムのエラーのほとんどは、誤った表現型に起因する可能性があります。
システム仕様に一致する88の関連のうち、44は全てのコホートで10−5 で有意ではなかったため、科学的な理由でGWAS Catalogから除外されていました。これらの多様体は、ノイズの多いデータセットが許容されるアプリケーションで依然として有用である可能性があるため、GWASkbに含めています。
別の36は、より重要な多様体と同じ座位(locus)にあったため除外されていました。ただし、これらは一般に完全連鎖不平衡(LD:Linkage Disequilibrium)ではなく、27はGWAS Catalogの多様体(LDLinkツールでr2 < 0.5)で弱いLDとされていました。何が重要な多様体であるかの判断は、科学的用途に依存する可能性があるため、私達はこれらの多様体をカタログ化することを主張します。
別の8つの多様体は、対象の研究と以前の研究の両方で有意であると判断されたため、含まれていませんでした。(GWAS Catalogガイドラインでは、そのような多様体をカタログ化する必要があることに注意してください)。
新規多様体と既存多様体間のLD
システムが見つけた新しい多様体を検証するために、多様体の機能を特徴付けることを目的とした一連の分析を実施しました。
まず、検出された多様体は(同じLDブロックに由来するため)既知の多様体またはそれらの間に存在する可能性があり、それにより、新しく検出された関連性の数が増えているのではないかと推論しました。
私達はLDをThousand Genomesデータセットから推定しました(補足ノート2を参照)。図3は、各新規多様体とGWAS Catalog内の最も近い多様体間のr2距離のヒストグラムを示しています。
r2スコアの分布は高度にマルチモーダルで、r2 = 1に大きなピークがありますが、r2 = 0には更に大きなピークがあります。
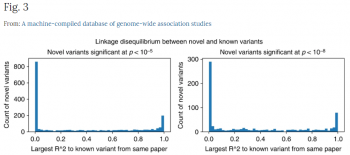
人間が手で作成した既存のキュレーションデータベースに存在しないGWASkb多様体とGWAS Catalogの多様体間の連鎖不平衡。Thousand Genomes datasetを使用して、多様体のペア間のr2を推定し、各GWASkb多様体と同じ論文で報告されている最も相関性の高いGWAS CatalogのSNPまでの距離を報告します。r2スコアの分布は高度にマルチモーダルです。多くのGWASkbバリアントは、GWAS CatalogのSNPと相関関係がありません(r2 = 0)。報告されたp値はカイ二乗検定で生成されています
r2> 0.5の閾値を使用して、新たに発見された[pmid、rsid、phen、pvalue]のセットをフィルタリングし、3170のペアを1494まで削減しました。手動でキュレーションされた既知のバリアントに近いLDの多様体を削除したのです。
削除した1676個の多様体のうち、765はThousand Genomes datasetに含まれていないか、最も近い既知の多様体もデータベースに含まれていませんでした。残りの911の一塩基多型(SNP:Single Nucleotide Polymorphism)は、既知の多様体と共にLDに含まれていました。
互いにLDに存在する新規多様体を排除する事により、関連性を更に1304に削減できました。従って、多くの亜種が既知の亜種とともにLDに含まれていますが、発見された亜種の40%以上は、以前にGWASデータベースで特定されている亜種にリンクしていないようです。
私達のシステムは同じLDブロックから複数の多様体を報告していますが、これらの多様体はまだ有用である可能性があります。これは、LDブロックが実際にどの多様体なのかわからない事と、LDブロックを定義するためのr2カットオフはいささか任意的であり、変化する可能性があるためです。
フィルタリングは、目標に応じてユーザーが実行する必要があると考えています。 これは、GWAS Centralリポジトリが採用しているアプローチでもあります。
有意性を推定するために他のアプローチで比較
2番目の分析は、新規に見つかった多様体の生物学的機能に焦点を当てています。神経変性疾患(ND:Neurodegenerative Diseases、自閉症、アルツハイマー、パーキンソンなどを含む27の特性)と自己免疫疾患(AI:autoimmune disorders、糖尿病、関節炎、ループスなどを含む23の特性)の2つの大きな表現型に焦点を当てています。
以下の分析では、GWASカタログまたはGWAS Centralの多様体を持つLDに存在しないバリアントのサブセットを考慮します。(283のND SNPおよび155のAI SNP)また、血液細胞だけでなく脳細胞でも高度に発現していることが判明した2セットの遺伝子を収集しました。具体的には、神経精神疾患と自己免疫疾患に関連するSNPは、脳と免疫細胞に関連する遺伝子の近くでより高度に濃縮されているはずと考えたためです。
実際、ND疾患に関連する多様体(合計32のND SNP)は、優先的脳発現を伴う遺伝子の200 kbp以内で有意に頻繁に発生し、AU特性に関連する多様体(合計15の多様体)は、 優先的血液発現(カイ二乗検定:p < 0.05、補足ノート2を参照)。
ただし、NDおよびAU多様体の大部分はコーディング領域から遠く離れて発見されたことに注意する必要があります。このSNPのセットも生物学的に意味をなすかどうかをテストするために、ゲノムの遺伝子間領域の多様体の機能に注釈を付けるツールであるGREATを使用しました。
GREATは、特に、遺伝子間領域を疾患オントロジー(DO:Disease Ontology)の用語とリンクし、特定の多様体セットに対して大幅に加筆された注釈を出力します。
ND SNPにGREATを適用すると、認知疾患(p < 10−32)、認知症(p < 10−23)、神経変性疾患(p < 10−23)など、ND関連の表現型で役割を果たすことが知られている領域で強い濃縮が見られました。
同様に、AI多様体はAI関連の用語と有意に関連しており、その中で最も重要なものは感染因子による疾患(p < 10−27)、ウイルス感染症(p < 10−19)、および自己免疫疾患(p < 10−17)。 実際、いずれかの多様体セットの上位20のDO用語はすべて、表現型の正しいファミリーに排他的に関連付けられていました。(補足表1および2)従って、私達が予測した多様体は外部ツールによる注釈と一致していました。
3.GWASkb:ゲノムワイド関連解析情報を論文から自動抽出(4/6)関連リンク
1)www.nature.com
A machine-compiled database of genome-wide association studies
2)github.com
kuleshov/gwaskb
3)gwaskb.stanford.edu
GWASKB


コメント