1.GWASkb:ゲノムワイド関連解析情報を論文から自動抽出(2/6)まとめ
・GWASkbは遺伝的多様体、表現型、およびp値を自動的に収集する
・構文解析、候補生成、候補分類の3段階を経て文書から関係性を抽出
・スタンフォードCoreNLPパイプラインやSnorkelを使って実現している
2.GWASkbの情報抽出方法
以下、www.nature.comより「A machine-compiled database of genome-wide association studies」の意訳です。元記事は2019年7月26日、Volodymyr Kuleshovさん、Jialin Dingさん、Christopher Voさん、Braden Hancockさん、Alexander Ratnerさん、Yang Liさん、Christopher Réさん、Serafim BatzoglouさんとMichael Snyderさんによる投稿です。
結果
GWASkbによる生物医学文献のキュレーションの自動化
簡単に言えば、私達は生物医学文献から遺伝子型と表現型の関係を抽出し、構造化されたデータベースに配置しました。(図1)
典型的な関連性は、遺伝的多様体(genetic variant)、それに関連する表現型、および関連性の重要性を示すp値から構成されます(補足ノート3を参照)。
GWASkbは、これら3つの特定の特性を収集します。関連性には、まだ私達のシステムがまだ収集処理をしていない追加のプロパティもあり、これらには、効果の大きさ、リスクアレル(risk allele:対立遺伝子)、目標母集団などが含まれます。最後に、どの出版物のどのページで見つけた情報かをPubmed IDで識別できるように、証拠として参照できるようにします。
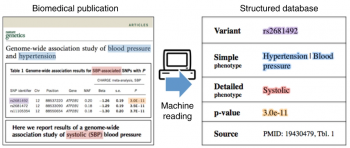
図1:GWASkbの編集に使用される自動情報抽出システムの概要図
GWASkbシステムは、入力としてPubMed Centralから取得した一連の生物医学出版物(左図)を受け取り、これらの出版物に記載されているGWASの関連性の構造化データベースを自動的に作成します(右)。各関連性について、システムは、遺伝的多様体(紫色)、高レベルの表現型(出版物に掲載されている全ての多様体に関連)、詳細な低レベルの表現型(利用可能な場合のみ、個々の多様体に固有な表現型、赤色)、およびp値(オレンジ)を識別します。頭字語も解釈されます(赤)
人間が手でキュレーションした表現型のデータベースは、非常に特定的である場合と(例:収縮期血圧が高い)、そうでない場合(例:心疾患)があります。GWASkbでは、シンプルで正確な表現型を報告します。前者は論文で扱っている全ての多様体に適用される高レベルな説明(例:炎症に対するタンパク質の影響)で、後者は特定の多様体に適用される詳細な説明(例:特定のタンパク質の名前)などです。
私達はまた、ユーザーがニーズに応じて調整できる大規模な関連性データセットを収集することも目指しています。このアプローチは、手動でキュレーションを行う際の一般的なアプローチである、信頼度の高い関係性のみを収集するアプローチよりも柔軟性があります。
情報抽出アルゴリズムを使用してGWASkbを作成する
GWASkbを生成するシステムは「遺伝的多様体(genetic variants)」、その「表現型(phenotypes)」、および「p値(p values)」という3つの重要な関係性を抽出するために、5つのコンポーネントに分割して実装されています。
システムの最初のコンポーネントは、全ての論文のタイトルと要約を解析して、そこから論文内の全ての多様体に関連付けられる単純な表現型を特定します。2番目のコンポーネントは、論文の本文を解析して、リファレンスSNPクラスターID(RSIDs:Reference SNP cluster ID)とそれに関連する正確な表現型の組み合わせを見つけます。
多くの場合、表現型は省略表記される(例:Body Mass IndexがBMIと省略されるなど)ため、3番目のコンポーネントはこれらの略語(例:BMI)を解決しようとします。
4番目のコンポーネントは、RSIDsとp値の組からp値を抽出します。最後に、5番目のコンポーネントは、これら全ての結果から単一の構造化データベースを構築します。
情報抽出システムのコンポーネントは、解析、候補生成、および分類の3つの段階で構成されています。(図2)
構文解析は、XMLや様々な形式のデータ(テキスト、構造データ、表、および/または視覚的手がかりを介して表現されるデータなど)に対応可能なナレッジベース構築フレームワークであるSnorkelを使用して実行されます。
コンテンツは最初に構造が解析されます。XMLツリーが走査され、テキストがテーブル、セル、段落、文などに割り当てられた階層データモデルに変換されます。
次に、文のトークン化、品詞タグ付け、構文解析を実行するスタンフォードCoreNLPパイプラインを使用して、各文またはセルの内容を解析します。
候補生成では、いくつかのターゲット関係(p値とRSIDのペアなど)への言及をテキストから識別します。正規表現または辞書が、有効な関係性を記述している可能性が高い候補を識別するために使用されます。(この段階で高精度を求めすぎると失敗します)。
最後に、分類段階で、機械学習分類器を使用して、これらの候補のどれが実際に正しい関係性を言及しているかかを判断します。
少数(4 – 12)の手動設計した特徴分類機能を備えたNaive Bayes分類器を使用し、最近提案されたデータプログラミングパラダイムを使用してモデルをトレーニングします。
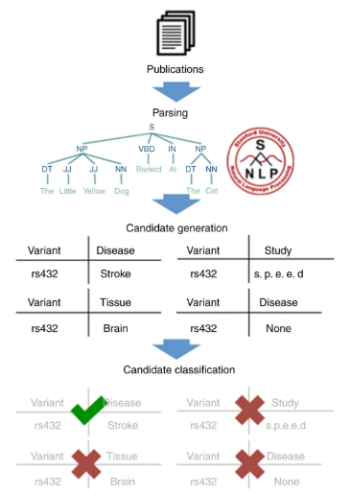
GWASkbシステムのモジュールの全体的な構造
システムには、多様体、表現型、p値を抽出し、頭字語を解決するための個別のモジュールが含まれています。各モジュールは3つのステージで構成されています。解析段階では、Stanford CoreNLPパイプラインを使用して論文を処理し、完全な構文解析を実行します。次に、ターゲットリレーション(たとえば、多様体と表現型)を指定すると、ターゲットリレーションが含まれている可能性があるリレーション候補の大規模なデータセットを生成します。次に、分類段階では、機械学習分類器を使用してどの候補が正しいかを判断します。
3.GWASkb:ゲノムワイド関連解析情報を論文から自動抽出(2/6)関連リンク
1)www.nature.com
A machine-compiled database of genome-wide association studies
2)github.com
kuleshov/gwaskb
3)gwaskb.stanford.edu
GWASKB


コメント