1.機械学習モデルの分類外データの検出を改良(2/3)まとめ
・Background Statisticsによって混乱が引き起こされるためOODの検出は失敗している可能性がある
・OOD検出手法はMNISTデータセットの画像をFashion-MNISTデータセットの画像と間違う事がある
・尤度計算は意味を持つ画素と背景画素の両方を含む、画像内のすべての画素から計算されてしまうため
2.Background Statisticsとは?
以下、ai.googleblog.comより「Improving Out-of-Distribution Detection in Machine Learning Models」の意訳です。元記事の投稿は2019年12月17日、Jie RenさんとBalaji Lakshminarayananさんによる投稿です。
密度モデルがOOD検出に失敗するのは何故でしょうか?
様々な手法を体系的に評価するために、現実世界で起こっている問題を真似る事にしました。
まず、公的に入手可能な NCBI catalogから入手したゲノム配列データを使用して、新しい細菌データセットを構築しました。
ゲノム配列データを模倣するために、ゲノム配列データを250塩基対の短い配列に断片化しました。250は現在のゲノム研究で一般的に取り扱われる長さです。その後、分類内データと分類外データに発見日を基準に区分けする事としました。データを分割する基準日前に発見された細菌を分類内データと定義し、その後に検出された細菌を分類外データと定義したのです。
次に、分類内データのゲノム配列に関して生成モデルをトレーニングし、尤度値をプロットすることで、モデルが分布内と分布外の入力をどれだけ区別出来ているかを調べました。
OODデータに対するの尤度のヒストグラムは、分類内データのヒストグラムとほぼ重複しており、生成モデルが2つの母集団からOODを区別できていない事を示しています。
同様の結果は、画像を扱う生成モデルの以前の研究で示されていました。例えば、Fashion-MNISTデータセット(衣服と履物の画像で構成される)の画像でトレーニングされたPixelCNN ++モデルは、MNISTデータセット(数字0-9の画像で構成される)、つまりOOD画像に対して高い可能性を割り当ててしまいます。
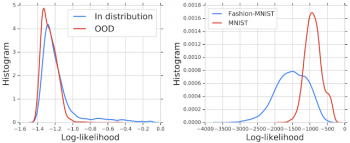
左図:分類内データおよび分類外データ(OOD)のゲノム配列の尤度値をヒストグラムにした図。分類内データとOODなゲノム配列を分離できていない可能性があります。
右図:Fashion-MNISTでトレーニングされ、MNISTで評価されたモデルの同様のプロット。このモデルは、分類内の画像よりもOOD(MNIST)に高い尤度値を割り当ててしまっています。
この失敗の原因を調査した時、背景画像の統計量(Background Statistics)によって混乱が引き起こされている可能性がある事がわかりました。
現象をより直感的に理解するには、入力が2つの部分、
(1)背景画像によって特徴付けられる「背景部分(background component)」
(2)分類内データに固有の意味を持つパターンによって特徴付けられる「意味を持つ部分(semantic component)」
で構成されると仮定しましょう。
たとえば、MNISTイメージは、背景部分と意味を持つ部分から構成されているとモデル化できます。人間が画像を解釈するとき、背景を無視するのは簡単で、主に意味を持つ情報、たとえば下の画像の「/」マークに集中できます。
しかし、尤度計算は意味を持つ画素と背景画素の両方を含む、画像内のすべての画素から計算されます。
意味を持つ画素のみを使用して尤度計算したいのですが、生の尤度は背景画像による大きな影響を受けます。

左上:Fashion-MNISTのサンプル画像。
左下:MNISTからのサンプル画像。
右:MNISTイメージの背景部分と意味を持つ部分
3.機械学習モデルの分類外データの検出を改良(2/3)関連リンク
1)ai.googleblog.com
Improving Out-of-Distribution Detection in Machine Learning Models
2)arxiv.org
Likelihood Ratios for Out-of-Distribution Detection
3)github.com
google-research/genomics_ood/


コメント