1.DeepVariant 1.0によるゲノム解析精度の向上(2/4)まとめ
・従来、ゲノム解析はIllumina社の機器の短いが正確な読み取りデータを利用してきた
・近年、Pacific BiosciencesやOxford Nanoporeなど長い読み取りが出来る機器が出現
・長い読み取りはエラーが多くなる可能性があるがDeepVariantは各機器でエラーを削減
2.DeepVariantが扱えるデータ
以下、ai.googleblog.comより「Improving the Accuracy of Genomic Analysis with DeepVariant 1.0」の意訳です。元記事の投稿は2020年9月18日、Andrew CarrollさんとPi-Chuan Changさんによる投稿です。
アイキャッチ画像のクレジットはPhoto by Nicolas Hoizey on Unsplash
ゲノム解析技術とDeepVariant
過去10年間、ゲノム配列データの大部分は、Illumina社の機器を使用して生成されてきました。この機器は、短い(75-250塩基)ですが正確な読み取りデータ(sequence data)を生成できました。
近年、より長い断片を読み取る事ができる新しい機器が利用可能になりました。これには、最大15,000塩基までの長く正確な読み取りデータを生成できるPacific Biosciencesや、最大100万塩基を読み取れますがエラー率が高くなるOxford Nanoporeなどがあります。研究者がどのタイプの読み取りデータを利用するかは、最終的な目的によって異なります。
DeepVariantはディープラーニングを使用しているため、これらの新しい機器に対してすばやく再トレーニングして、非常に正確に読み取りデータを識別できます。
バリアントコール(variant call、解析対象の塩基配列中のどの位置にどのようなvariantsが発生しているかを特定する事)の見逃しは、遺伝的障害の原因となったvariantsを見逃す事を意味する可能性があるため、正確さが重要ですが、その一方、バリアントコールの誤検知は、間違ったvariantsを識別してしまう可能性があります。
従来の最先端の手法は、35-fold coverageのIlluminaの全ゲノムで約99.1%の精度(約73,000エラー)を達成する事が出来ましたが、DeepVariantの初期バージョン(v0.10)の精度は99.4%(約46,000エラー)で、これは38%のエラー削減に相当します。
DeepVariant v1.0は、前回のDeepVariantリリース(v0.10)と比較して、Illuminaのエラーをさらに約22%、PacBioのエラーをさらに約52%削減しました。
DeepVariantの概要
DeepVariantは、遺伝的変異を識別するタスクを画像分類問題として扱う畳み込みニューラルネットワーク(CNN)です。DeepVariantは、基本的にはマルチチャネルイメージをテンソル(tensors)として構築します。
ここで、各チャネルは読み取りデータの一定の側面を表現しています。
例えば、
・ゲノム配列の読み取りデータ(read base)
・異なる読み取りデータ間の整列品質(mapping quality)
・特定の読み取りデータが代替対立遺伝子をサポートするかどうか(read supports variant)
などです。
次に、これらのデータを分析し、推測したゲノムタイプを3種の数字(0、1、または2)で出力します。この数字は、特定の代替対立遺伝子(alternate allele)のコピーが幾つ存在するかに対応します。
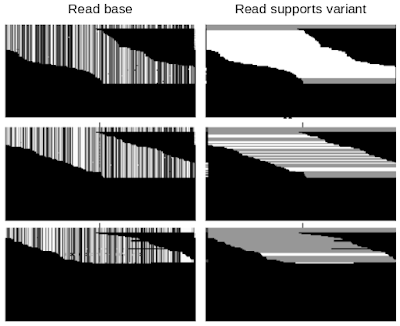
DeepVariantデータの例
各パネルの各行の画素は、単一の読み取りデータ、つまり短いゲノム配列に対応します。上段、中段、下段でDeepVariantは異なる数字を出力しています。6つのデータチャネルのうち以下の2つだけが表示されています。
read base:画素値はそれぞれ4つのベース(A、C、G、またはT)にマッピングされます。
Read supports variant:白は読み取りが特定の対立遺伝子と一致していることを意味し、灰色はそうではない事を意味します。
上段:DeepVariantによって「2」として分類されたデータです。これは、両方の染色体がvariantsの対立遺伝子と一致することを意味します。
中段:「1」として分類されたデータです。1つの染色体がvariantsの対立遺伝子と一致することを意味します。
下段:「0」として分類されたデータです。variantsの対立遺伝子が両方の染色体から欠落していることを意味します。
3.DeepVariant 1.0によるゲノム解析精度の向上(2/4)関連リンク
1)ai.googleblog.com
Improving the Accuracy of Genomic Analysis with DeepVariant 1.0
2)www.nature.com
A universal SNP and small-indel variant caller using deep neural networks
3)github.com
google / deepvariant
kishwarshafin / pepper
4)www.nist.gov
Genome in a Bottle