1.ProtENN:ディープラーニングでタンパク質に注釈付けをする(3/3)まとめ
・ProtENNが配列並びベースの手法と相補的な情報を学習することを実証してアンサンブルした
・取り組みの成果として680万件の新しいタンパク質配列の注釈セットであるPfam-Nを公開
・従来の解釈可能な手法を採用してニューラルネットが着目した場所を特定できるようになった
2.ProtCNNの性能
以下、ai.googleblog.comより「Using Deep Learning to Annotate the Protein Universe」の意訳です。元記事は2022年3月2日、Maxwell BileschiさんとLucy Colwellさんによる投稿です。
アイキャッチ画像のクレジットはPhoto by LyfeFuel on Unsplash
2番目の評価では、ランダムに分割した学習用データセットとテストセットを使用し、分類がどの程度困難であるかの推定に基づいてサンプルを分割します。これらの困難さの尺度には以下のものがあります。
(1)テストサンプル例と最も近い学習サンプルの類似度
(2)真実のクラスに属する学習サンプルの数(ほんの一握りの学習サンプルしかない際に機能を正確に予測することはより困難です)
私達の研究の背景を説明するために、最も広く使われているモデルと評価環境で性能を評価しました。特に以下のモデルを比較対象として評価しました。
(1)BLAST:配列の並びを利用して距離を測定し、機能を推測する最近傍法モデル
(2)プロファイル隠れマルコフモデル(TPHMM、phmmer:profile hidden Markov models)
それぞれについて、上述の配列並びの類似度に基づいてモデル性能を評価する事を含みます。これらの比較対象モデルとProtCNNおよびCNNのアンサンブルであるProtENNを比較しました。
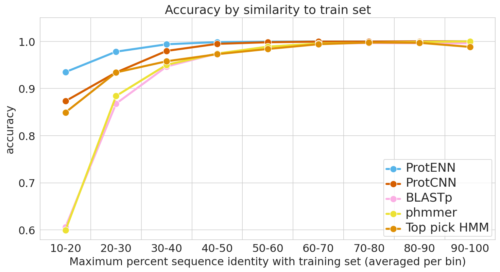
各モデルの汎化能力を、最も難しい例(左)から最も簡単な例(右)まで測定しています。
再現性と解釈可能な結果
また、欧州分子生物学研究所欧州バイオインフォマティクス研究所(EMBL-EBI)の国際的に著名な専門家であるPfamチームと共同で、私達の方法論的概念実証が実際の配列に適用できるかどうかを検証しました。
ProtENNが配列並びベースの手法と相補的な情報を学習することを実証し、2つのアプローチのアンサンブルを作成し、どちらかの手法が単独で行うよりも多くの配列のラベル付けができることを示しました。この取り組みの成果として、680万件の新しいタンパク質配列の注釈セットであるPfam-Nを一般に公開しました。
これらの手法と分類タスクとしての成功を見た後、私達はこれらのネットワークを検査し、embeddingsが一般的に有用であるかどうかを理解しようとしました。
私達は、モデル予測、embeddings、入力配列の関係を探索できるツールを構築し、ユーザ操作可能なWebページとして公開しました。その結果、embeddings空間において類似配列がクラスタリングされていることが分かりました。
さらに、私達が選択した拡張CNN(dilated CNN)というネットワークアーキテクチャにより、クラスアクティベーションマッピング(CAM:Class Activation Mapping)や十分入力サブセット(SIS:Sufficient Input Subsets、他の全ての入力特徴が欠落していても、同じ決定に到達するのに十分な最小の特徴のサブセットを識別する手法)といったこれまでに発見された解釈可能な手法を採用して、ニューラルネットワークの予測に関わる部分配列を特定することが可能となりました。
このアプローチにより、私達のネットワークは配列の汎用的な関連要素に着目し、その機能を予測している事がわかりました。
まとめと今後の課題
私たちは、ここ数年、タンパク質の構造と機能の理解にMLを適用することで見られる進歩に興奮しています。
この進歩は、AlphaFoldやCAFAをはじめ、多くのワークショップや学会での研究発表など、より広い研究コミュニティからの貢献が反映されています。私たちは、この成果を基に、専門知識やデータを共有した研究者たちとの共同作業を継続し、さらにMLを発展させることで、タンパク質の世界をさらに明らかにすることができると考えています。
謝辞
原稿の共著者の皆さんに感謝します。
Maysam Moussalem, Jamie Smith, Eli Bixby, Babak Alipanahi, Shanqing Cai, Cory McLean, Abhinay Ramparasad, Steven Kearnes, Zack Nado, そして Tom Small。また、Pfam-Nの公開にご協力いただいたEMBL-EBIのPfamチームにも感謝いたします。
3.ProtENN:ディープラーニングでタンパク質に注釈付けをする(3/3)関連リンク
1)ai.googleblog.com
Using Deep Learning to Annotate the Protein Universe
2)www.nature.com
Using deep learning to annotate the protein universe
3)github.com
google-research/using_dl_to_annotate_protein_universe/