1.ProtENN:ディープラーニングでタンパク質に注釈付けをする(1/3)まとめ
・タンパク質のアミノ酸配列の構造と機能の関係を理解することは科学にとって非常に重要
・機能が未知のタンパク質はまだ多く、信頼性の高い注釈が付与されていないものも多い
・ProtENNはタンパク質機能注釈セットに約680万項目を追加することを可能にしたモデル
2.ProtENNとは?
以下、ai.googleblog.comより「Using Deep Learning to Annotate the Protein Universe」の意訳です。元記事は2022年3月2日、Maxwell BileschiさんとLucy Colwellさんによる投稿です。
飲む方のプロテインを意識したアイキャッチ画像のクレジットはPhoto by LyfeFuel on Unsplash
タンパク質は、すべての生物に存在する必須分子です。私たちの身体の構造と機能において中心的な役割を果たしており、薬から洗濯用洗剤などの日用品に至るまで、私たちが毎日出会う多くの製品にも含まれています。
タンパク質はアミノ酸が連なったものです。犬と猫が異なるように、タンパク質にも複数の構成要素があり、これらをタンパク質ドメイン(protein domains)と呼びます。タンパク質のアミノ酸配列、つまりドメインと、その構造や機能の関係を理解することは、科学的に非常に重要な意味を持つ長年の課題です。

大腸菌由来のTrpCFという構造がわかっているタンパク質の例
機能予測モデルで使用されている部分が緑色でハイライトされています。
このタンパク質は、人の食事に欠かせないトリプトファンを生成します。
近年、DeepMind社のAlphaFoldに見られるように、アミノ酸配列からタンパク質の構造を計算して予測する技術が進歩していることは、多くの人が知っていることと思います。
同様に、科学界には、アミノ酸配列からタンパク質の機能を直接推測するために計算ツールを使用してきた長い歴史があります。例えば、広く使われているタンパク質群用データベースPfamには、タンパク質ドメインの機能を説明する非常に詳細な計算アノテーションが多数含まれています。(例:グロビン・ファミリー、トリプシン・ファミリーなど)。
既存手法では何億ものタンパク質の機能を予測することに成功していますが、機能が未知のタンパク質はまだ多く、例えば、微生物タンパク質の少なくとも3分の1は信頼性の高い注釈が付与されていません。公開データベースに登録されたタンパク質配列の量と多様性が急速に増加し続ける中、分岐の多い配列に対して正確に機能を予測することがますます重要になってきています。
Nature Biotechnologyに掲載された「Using Deep Learning to Annotate the Protein Universe」では、私達はタンパク質の機能を確実に予測する機械学習(ML:Machine Learning)技術について述べています。この手法はProtENNと呼ばれ、Pfamの有名で信頼できるタンパク質機能注釈セットに約680万項目を追加することを可能にしたものです。これは、過去10年間の進歩の合計にほぼ等しく、Pfam-Nとしてリリースしています。
この方向でさらに研究を進めるために、ProtENNモデルと、研究者が私達の技術を試すことができる論文サイトdistill.pubのような対話型記事を公開しています。この対話型ツールは、ユーザーが配列を入力し、ブラウザ上でリアルタイムに、設定不要でタンパク質の機能を予測した結果を得ることができるものです。この記事では、この成果の概要と、タンパク質の世界のより詳細な解明に向けてどのように前進しているかを紹介します。
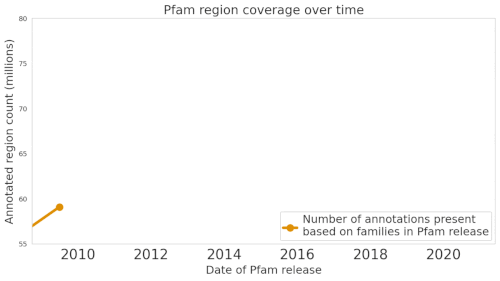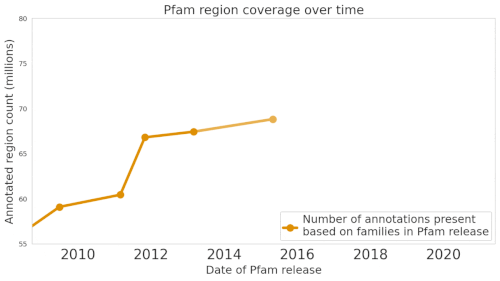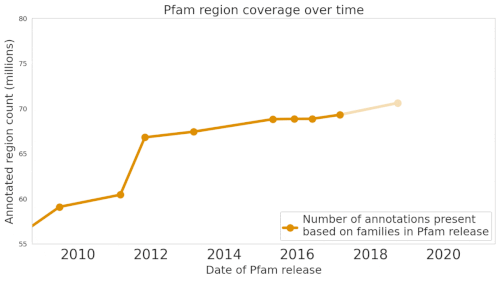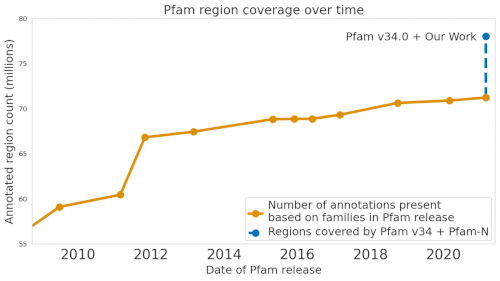
Pfamデータベースは、タンパク質群とその配列の大規模なコレクションです。
私たちのMLモデルProtENNは、このデータベースの680万以上のタンパク質ドメインの注釈付けに貢献しました
3.ProtENN:ディープラーニングでタンパク質に注釈付けをする(1/3)関連リンク
1)ai.googleblog.com
Using Deep Learning to Annotate the Protein Universe
2)www.nature.com
Using deep learning to annotate the protein universe
3)github.com
google-research/using_dl_to_annotate_protein_universe/